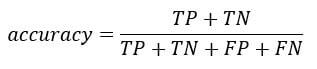Árvore de decisão em R: árvore de classificação com exemplo
O que são árvores de decisão?
Árvores de decisão são algoritmos versáteis de aprendizado de máquina que podem executar tarefas de classificação e regressão. São algoritmos muito poderosos, capazes de ajustar conjuntos de dados complexos. Além disso, as árvores de decisão são componentes fundamentais das florestas aleatórias, que estão entre os algoritmos de aprendizado de máquina mais potentes disponíveis atualmente.
Treinamento e visualização de árvores de decisão em R
Para construir sua primeira árvore de decisão no exemplo R, procederemos da seguinte forma neste tutorial de árvore de decisão:
- Etapa 1: importe os dados
- Etapa 2: limpe o conjunto de dados
- Etapa 3: criar conjunto de treinamento/teste
- Etapa 4: construir o modelo
- Etapa 5: faça previsões
- Etapa 6: Avalie o desempenho
- Etapa 7: ajuste os hiperparâmetros
Etapa 1) Importe os dados
Se você está curioso sobre o destino do Titanic, pode assistir a este vídeo no Youtube. O objetivo deste conjunto de dados é prever quais pessoas têm maior probabilidade de sobreviver após a colisão com o iceberg. O conjunto de dados contém 13 variáveis e 1309 observações. O conjunto de dados é ordenado pela variável X.
set.seed(678) path <- 'https://raw.githubusercontent.com/guru99-edu/R-Programming/master/titanic_data.csv' titanic <-read.csv(path) head(titanic)
Saída:
## X pclass survived name sex ## 1 1 1 1 Allen, Miss. Elisabeth Walton female ## 2 2 1 1 Allison, Master. Hudson Trevor male ## 3 3 1 0 Allison, Miss. Helen Loraine female ## 4 4 1 0 Allison, Mr. Hudson Joshua Creighton male ## 5 5 1 0 Allison, Mrs. Hudson J C (Bessie Waldo Daniels) female ## 6 6 1 1 Anderson, Mr. Harry male ## age sibsp parch ticket fare cabin embarked ## 1 29.0000 0 0 24160 211.3375 B5 S ## 2 0.9167 1 2 113781 151.5500 C22 C26 S ## 3 2.0000 1 2 113781 151.5500 C22 C26 S ## 4 30.0000 1 2 113781 151.5500 C22 C26 S ## 5 25.0000 1 2 113781 151.5500 C22 C26 S ## 6 48.0000 0 0 19952 26.5500 E12 S ## home.dest ## 1 St Louis, MO ## 2 Montreal, PQ / Chesterville, ON ## 3 Montreal, PQ / Chesterville, ON ## 4 Montreal, PQ / Chesterville, ON ## 5 Montreal, PQ / Chesterville, ON ## 6 New York, NY
tail(titanic)
Saída:
## X pclass survived name sex age sibsp ## 1304 1304 3 0 Yousseff, Mr. Gerious male NA 0 ## 1305 1305 3 0 Zabour, Miss. Hileni female 14.5 1 ## 1306 1306 3 0 Zabour, Miss. Thamine female NA 1 ## 1307 1307 3 0 Zakarian, Mr. Mapriededer male 26.5 0 ## 1308 1308 3 0 Zakarian, Mr. Ortin male 27.0 0 ## 1309 1309 3 0 Zimmerman, Mr. Leo male 29.0 0 ## parch ticket fare cabin embarked home.dest ## 1304 0 2627 14.4583 C ## 1305 0 2665 14.4542 C ## 1306 0 2665 14.4542 C ## 1307 0 2656 7.2250 C ## 1308 0 2670 7.2250 C ## 1309 0 315082 7.8750 S
Na saída head e tail, você pode notar que os dados não são embaralhados. Este é um grande problema! Ao dividir seus dados entre um conjunto de treinamento e um conjunto de teste, você selecionará só o passageiro das classes 1 e 2 (nenhum passageiro da classe 3 está entre os 80% melhores das observações), o que significa que o algoritmo nunca verá as características do passageiro da classe 3. Esse erro levará a uma previsão ruim.
Para superar esse problema, você pode usar a função sample().
shuffle_index <- sample(1:nrow(titanic)) head(shuffle_index)
Explicação do código R da árvore de decisão
- sample(1:nrow(titanic)): Gera uma lista aleatória de índices de 1 a 1309 (ou seja, o número máximo de linhas).
Saída:
## [1] 288 874 1078 633 887 992
Você usará este índice para embaralhar o conjunto de dados do Titanic.
titanic <- titanic[shuffle_index, ] head(titanic)
Saída:
## X pclass survived ## 288 288 1 0 ## 874 874 3 0 ## 1078 1078 3 1 ## 633 633 3 0 ## 887 887 3 1 ## 992 992 3 1 ## name sex age ## 288 Sutton, Mr. Frederick male 61 ## 874 Humblen, Mr. Adolf Mathias Nicolai Olsen male 42 ## 1078 O'Driscoll, Miss. Bridget female NA ## 633 Andersson, Mrs. Anders Johan (Alfrida Konstantia Brogren) female 39 ## 887 Jermyn, Miss. Annie female NA ## 992 Mamee, Mr. Hanna male NA ## sibsp parch ticket fare cabin embarked home.dest## 288 0 0 36963 32.3208 D50 S Haddenfield, NJ ## 874 0 0 348121 7.6500 F G63 S ## 1078 0 0 14311 7.7500 Q ## 633 1 5 347082 31.2750 S Sweden Winnipeg, MN ## 887 0 0 14313 7.7500 Q ## 992 0 0 2677 7.2292 C
Etapa 2) Limpe o conjunto de dados
A estrutura dos dados mostra que algumas variáveis possuem NAs. A limpeza de dados deve ser feita da seguinte forma
- Solte as variáveis home.dest, cabine, nome, X e ticket
- Crie variáveis de fator para pclass e sobreviveu
- Abandone o NA
library(dplyr)
# Drop variables
clean_titanic <- titanic % > %
select(-c(home.dest, cabin, name, X, ticket)) % > %
#Convert to factor level
mutate(pclass = factor(pclass, levels = c(1, 2, 3), labels = c('Upper', 'Middle', 'Lower')),
survived = factor(survived, levels = c(0, 1), labels = c('No', 'Yes'))) % > %
na.omit()
glimpse(clean_titanic)
Explicação do código
- select(-c(home.dest, cabine, nome, X, ticket)): Elimina variáveis desnecessárias
- pclass = factor (pclass, níveis = c (1,2,3), rótulos = c ('Upper', 'Middle', 'Lower')): Adicione rótulo à variável pclass. 1 torna-se Superior, 2 torna-se Médio e 3 torna-se inferior
- factor(sobreviveu, níveis = c(0,1), rótulos = c('Não', 'Sim')): Adiciona rótulo à variável sobreviveu. 1 torna-se Não e 2 torna-se Sim
- na.omit(): Remove as observações de NA
Saída:
## Observations: 1,045 ## Variables: 8 ## $ pclass <fctr> Upper, Lower, Lower, Upper, Middle, Upper, Middle, U... ## $ survived <fctr> No, No, No, Yes, No, Yes, Yes, No, No, No, No, No, Y... ## $ sex <fctr> male, male, female, female, male, male, female, male... ## $ age <dbl> 61.0, 42.0, 39.0, 49.0, 29.0, 37.0, 20.0, 54.0, 2.0, ... ## $ sibsp <int> 0, 0, 1, 0, 0, 1, 0, 0, 4, 0, 0, 1, 1, 0, 0, 0, 1, 1,... ## $ parch <int> 0, 0, 5, 0, 0, 1, 0, 1, 1, 0, 0, 1, 1, 0, 2, 0, 4, 0,... ## $ fare <dbl> 32.3208, 7.6500, 31.2750, 25.9292, 10.5000, 52.5542, ... ## $ embarked <fctr> S, S, S, S, S, S, S, S, S, C, S, S, S, Q, C, S, S, C...
Etapa 3) Criar conjunto de treinamento/teste
Antes de treinar seu modelo, você precisa realizar duas etapas:
- Crie um conjunto de treinamento e teste: você treina o modelo no conjunto de treinamento e testa a previsão no conjunto de teste (ou seja, dados não vistos)
- Instale rpart.plot do console
A prática comum é dividir os dados 80/20, 80% dos dados servem para treinar o modelo e 20% para fazer previsões. Você precisa criar dois quadros de dados separados. Você não quer mexer no conjunto de testes até terminar de construir seu modelo. Você pode criar um nome de função create_train_test() que receba três argumentos.
create_train_test(df, size = 0.8, train = TRUE) arguments: -df: Dataset used to train the model. -size: Size of the split. By default, 0.8. Numerical value -train: If set to `TRUE`, the function creates the train set, otherwise the test set. Default value sets to `TRUE`. Boolean value.You need to add a Boolean parameter because R does not allow to return two data frames simultaneously.
create_train_test <- function(data, size = 0.8, train = TRUE) {
n_row = nrow(data)
total_row = size * n_row
train_sample < - 1: total_row
if (train == TRUE) {
return (data[train_sample, ])
} else {
return (data[-train_sample, ])
}
}
Explicação do código
- function(data, size=0.8, train = TRUE): Adicione os argumentos na função
- n_row = nrow(data): conta o número de linhas no conjunto de dados
- total_row = size*n_row: Retorna a enésima linha para construir o conjunto de trens
- train_sample <- 1:total_row: Selecione a primeira linha até a enésima linha
- if (train ==TRUE){ } else { }: Se a condição for definida como verdadeira, retorne o conjunto de trem, caso contrário, o conjunto de teste.
Você pode testar sua função e verificar a dimensão.
data_train <- create_train_test(clean_titanic, 0.8, train = TRUE) data_test <- create_train_test(clean_titanic, 0.8, train = FALSE) dim(data_train)
Saída:
## [1] 836 8
dim(data_test)
Saída:
## [1] 209 8
O conjunto de dados de treinamento possui 1046 linhas, enquanto o conjunto de dados de teste possui 262 linhas.
Você usa a função prop.table() combinada com table() para verificar se o processo de randomização está correto.
prop.table(table(data_train$survived))
Saída:
## ## No Yes ## 0.5944976 0.4055024
prop.table(table(data_test$survived))
Saída:
## ## No Yes ## 0.5789474 0.4210526
Em ambos os conjuntos de dados, a quantidade de sobreviventes é a mesma, cerca de 40%.
Instale rpart.plot
rpart.plot não está disponível nas bibliotecas conda. Você pode instalá-lo a partir do console:
install.packages("rpart.plot")
Etapa 4) Construa o modelo
Você está pronto para construir o modelo. A sintaxe para a função da árvore de decisão Rpart é:
rpart(formula, data=, method='') arguments: - formula: The function to predict - data: Specifies the data frame- method: - "class" for a classification tree - "anova" for a regression tree
Você usa o método de classe porque prevê uma classe.
library(rpart) library(rpart.plot) fit <- rpart(survived~., data = data_train, method = 'class') rpart.plot(fit, extra = 106
Explicação do código
- rpart(): Função para ajustar o modelo. Os argumentos são:
- sobreviveu ~.: Fórmula das Árvores de Decisão
- dados = data_train: conjunto de dados
- método = 'classe': ajusta um modelo binário
- rpart.plot(fit, extra= 106): Trace a árvore. Os recursos extras são definidos como 101 para exibir a probabilidade da 2ª classe (útil para respostas binárias). Você pode consultar o vinheta para obter mais informações sobre as outras opções.
Saída:
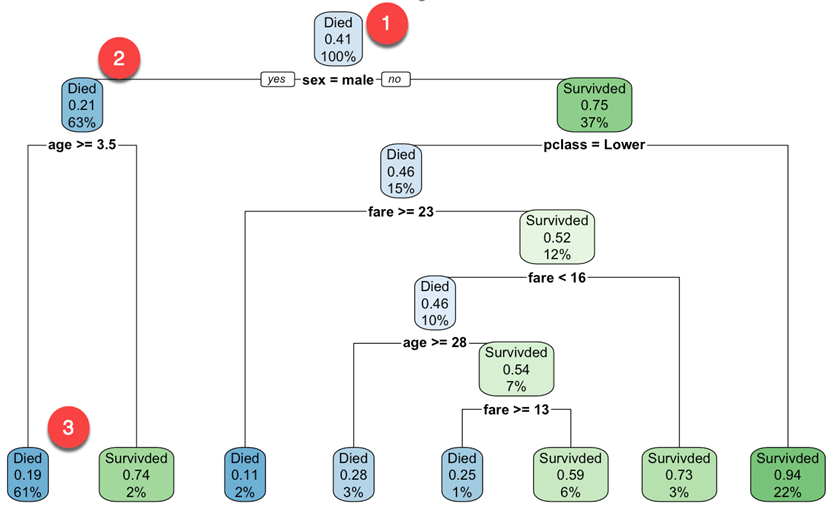
Você começa no nó raiz (profundidade 0 sobre 3, parte superior do gráfico):
- No topo, está a probabilidade geral de sobrevivência. Mostra a proporção de passageiros que sobreviveram ao acidente. 41 por cento dos passageiros sobreviveram.
- Este nó pergunta se o sexo do passageiro é masculino. Se sim, então você desce até o nó filho esquerdo da raiz (profundidade 2). 63 por cento são homens com uma probabilidade de sobrevivência de 21 por cento.
- No segundo nó, você pergunta se o passageiro do sexo masculino tem mais de 3.5 anos. Se sim, então a chance de sobrevivência é de 19%.
- Você continua assim para entender quais características afetam a probabilidade de sobrevivência.
Observe que uma das muitas qualidades das árvores de decisão é que elas exigem muito pouca preparação de dados. Em particular, eles não exigem dimensionamento ou centralização de recursos.
Por padrão, a função rpart() usa o Gini medida de impureza para dividir a nota. Quanto maior o coeficiente de Gini, mais instâncias diferentes dentro do nó.
Etapa 5) Faça uma previsão
Você pode prever seu conjunto de dados de teste. Para fazer uma previsão, você pode usar a função prever(). A sintaxe básica da previsão para a árvore de decisão R é:
predict(fitted_model, df, type = 'class')
arguments:
- fitted_model: This is the object stored after model estimation.
- df: Data frame used to make the prediction
- type: Type of prediction
- 'class': for classification
- 'prob': to compute the probability of each class
- 'vector': Predict the mean response at the node level
Você deseja prever quais passageiros têm maior probabilidade de sobreviver após a colisão no conjunto de teste. Isso significa que você saberá entre esses 209 passageiros qual sobreviverá ou não.
predict_unseen <-predict(fit, data_test, type = 'class')
Explicação do código
- prever(fit, data_test, type = 'class'): Prever a classe (0/1) do conjunto de teste
Testando o passageiro que não conseguiu e os que conseguiram.
table_mat <- table(data_test$survived, predict_unseen) table_mat
Explicação do código
- table(data_test$survived, predict_unseen): Crie uma tabela para contar quantos passageiros são classificados como sobreviventes e faleceram em comparação com a classificação correta da árvore de decisão em R
Saída:
## predict_unseen ## No Yes ## No 106 15 ## Yes 30 58
O modelo previu corretamente 106 passageiros mortos, mas classificou 15 sobreviventes como mortos. Por analogia, o modelo classificou erroneamente 30 passageiros como sobreviventes, mas eles estavam mortos.
Etapa 6) Medir o desempenho
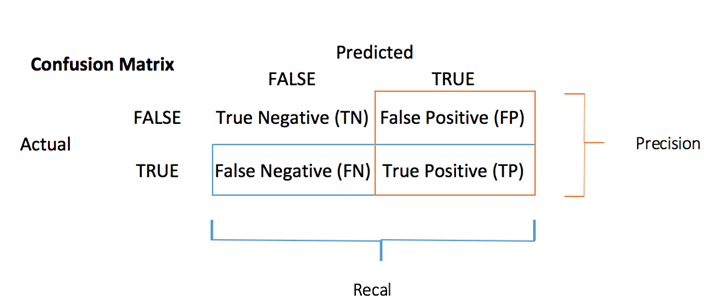
Você pode calcular uma medida de precisão para tarefa de classificação com o matriz de confusão:
O matriz de confusão é a melhor escolha para avaliar o desempenho da classificação. A ideia geral é contar o número de vezes que instâncias Verdadeiras são classificadas como Falsas.
Cada linha em uma matriz de confusão representa um alvo real, enquanto cada coluna representa um alvo previsto. A primeira linha desta matriz considera passageiros mortos (a classe False): 106 foram corretamente classificados como mortos (Verdadeiro negativo), enquanto o restante foi erroneamente classificado como sobrevivente (Falso positivo). A segunda linha considera os sobreviventes, a classe positiva foi 58 (Verdadeiro-positivo), Enquanto que o Verdadeiro negativo foi 30.
Você pode calcular o teste de precisão da matriz de confusão:
É a proporção de verdadeiros positivos e verdadeiros negativos sobre a soma da matriz. Com R, você pode codificar da seguinte forma:
accuracy_Test <- sum(diag(table_mat)) / sum(table_mat)
Explicação do código
- sum(diag(table_mat)): Soma da diagonal
- sum(table_mat): Soma da matriz.
Você pode imprimir a precisão do conjunto de teste:
print(paste('Accuracy for test', accuracy_Test))
Saída:
## [1] "Accuracy for test 0.784688995215311"
Você tem uma pontuação de 78% para o conjunto de testes. Você pode replicar o mesmo exercício com o conjunto de dados de treinamento.
Etapa 7) Ajuste os hiperparâmetros
A árvore de decisão em R possui vários parâmetros que controlam aspectos do ajuste. Na biblioteca de árvore de decisão rpart, você pode controlar os parâmetros usando a função rpart.control(). No código a seguir, você introduz os parâmetros que irá ajustar. Você pode consultar o vinheta para outros parâmetros.
rpart.control(minsplit = 20, minbucket = round(minsplit/3), maxdepth = 30) Arguments: -minsplit: Set the minimum number of observations in the node before the algorithm perform a split -minbucket: Set the minimum number of observations in the final note i.e. the leaf -maxdepth: Set the maximum depth of any node of the final tree. The root node is treated a depth 0
Procederemos da seguinte forma:
- Construir função para retornar precisão
- Ajuste a profundidade máxima
- Ajuste o número mínimo de amostras que um nó deve ter antes de poder ser dividido
- Ajuste o número mínimo de amostras que um nó folha deve ter
Você pode escrever uma função para exibir a precisão. Você simplesmente agrupa o código que usou antes:
- prever: prever_unseen <- prever (ajuste, teste de dados, tipo = 'classe')
- Produzir tabela: table_mat <- table(data_test$survived, predict_unseen)
- Precisão do cálculo: precisão_Test <- sum(diag(table_mat))/sum(table_mat)
accuracy_tune <- function(fit) {
predict_unseen <- predict(fit, data_test, type = 'class')
table_mat <- table(data_test$survived, predict_unseen)
accuracy_Test <- sum(diag(table_mat)) / sum(table_mat)
accuracy_Test
}
Você pode tentar ajustar os parâmetros e ver se consegue melhorar o modelo em relação ao valor padrão. Como lembrete, você precisa obter uma precisão superior a 0.78
control <- rpart.control(minsplit = 4,
minbucket = round(5 / 3),
maxdepth = 3,
cp = 0)
tune_fit <- rpart(survived~., data = data_train, method = 'class', control = control)
accuracy_tune(tune_fit)
Saída:
## [1] 0.7990431
Com o seguinte parâmetro:
minsplit = 4 minbucket= round(5/3) maxdepth = 3cp=0
Você obtém um desempenho superior ao modelo anterior. Parabéns!
Resumo
Podemos resumir as funções para treinar um algoritmo de árvore de decisão em R
| Biblioteca | Objetivo | função | Aula | Parâmetros Técnicos | Detalhes |
|---|---|---|---|---|---|
| parte | Árvore de classificação de trens em R | parte() | classe | fórmula, df, método | |
| parte | Treinar árvore de regressão | parte() | Anova | fórmula, df, método | |
| parte | Trace as árvores | rpart.plot() | modelo ajustado | ||
| base | predizer | prever() | classe | modelo equipado, tipo | |
| base | predizer | prever() | prov | modelo equipado, tipo | |
| base | predizer | prever() | vetor | modelo equipado, tipo | |
| parte | parâmetros de controle | rpart.control() | divisão mínima | Defina o número mínimo de observações no nó antes que o algoritmo execute uma divisão | |
| minbucket | Defina o número mínimo de observações na nota final, ou seja, na folha | ||||
| profundidade máxima | Defina a profundidade máxima de qualquer nó da árvore final. O nó raiz é tratado com profundidade 0 | ||||
| parte | Modelo de trem com parâmetro de controle | parte() | fórmula, df, método, controle |
Nota: Treine o modelo em dados de treinamento e teste o desempenho em um conjunto de dados não visto, ou seja, conjunto de teste.